Laboratoire de biologie moléculaire
Date de mise à jour : 22 août 2023
Principales Activités
Grâce à la synergie avec le laboratoire de microbiologie, nous pouvons caractériser les flores microbiennes cultivables isolées de matrices alimentaires variées (céréales, fruits, produits laitiers, aliments fermentés …) par une approche de barcoding. Cette approche consiste, après isolement de souches bactériennes ou fongiques, à séquencer par méthode Sanger une région spécifique amplifiée par PCR qui permettra son identification. L’identification moléculaire des souches microbiennes issues d’isolats, est complémentée par des études morphologiques macro et microscopiques réalisées au laboratoire de microbiologie.
Les communautés microbiennes associées aux aliments, y compris non cultivables, peuvent également être analysées et caractérisées par des méthodes sans a priori telles que le métabarcoding mis en œuvre en routine au laboratoire de biologie moléculaire. Cette approche permet de décrire la composition taxonomique des communautés étudiées grâce aux méthodes de séquençage de nouvelle génération (NGS) aussi bien en short reads (Illumina ) qu’en long reads (Oxford Nanopores, MinION).
Montpellier
L’activité principale développée est la caractérisation des communautés microbiennes,bactériennes et fongiques, cultivables et non cultivables, par méthodes avec ou sans a priori) associées aux aliments. Cette activité s’articule autour de différentes approches :
- Métabarcoding pour la caractérisation des communautés bactériennes et/ou fongiques. Cette approche s’appuie sur les outils de séquençage haut-débit (NGS) et nécessite l’amplification de marqueur moléculaires (16S, ITS, …) par PCR à partir de l’ADN extrait
- Métagénomique shot-gun, qui consiste à séquencer directement l’ADN métagénomique extrait sans amplification
- Transcriptomique
- PCR quantitative
Expertise bio-informatique et programmation (R, Python) pour le traitement des données métagénomiques et metabarcoding
La Réunion
Le laboratoire de biologie moléculaire à la Réunion permet d’extraire et d’amplifier l’ADN mais aussi de séparer l’ADN par la technique DGGE.
Principaux Équipements
Montpellier
Les équipements associés sont localisés dans les laboratoires 119 et 138, au premier étage de la maison de la technologie (bâtiment 16). Le laboratoire s’appuie également sur des collaborations avec le grand plateau technique régional de génotypage (https://www.gptr-lr-genotypage.com/) et le plateau de génotypage-Séquençage Genseq (https://www.gptr-lr-genotypage.com/plateaux-techniques/plateau-genseq-um/presentation) pour le séquençage Illumina ainsi que sur le plateau de PCR quantitative haut-débit situé à l’Université de Montpellier, tous trois membres de la plateforme Montpellier GenomiX (MGX, https://www.mgx.cnrs.fr/).
- Hotte chimique, Sorbonne, bains secs et centrifugeuses pour l’extraction des acides nucléiques.
- Nanodrop, Qubit pour le dosage des acides nucléiques.
- Hotte de pipetage pour la préparation de mix PCR.
- Thermocycleurs (lister les marques et modèles ?) pour amplifications par PCR.
- Cuves à électrophorèse et système de prise d’image pour gels d’agarose.
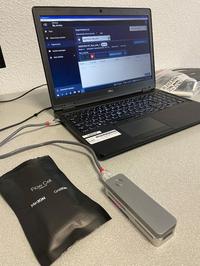
- Séquenceur Oxford Nanopores technologies MinION Mk1b.
La Réunion
- Appareillage DGGE
- FastPrep pour le broyage des échantillons
- Incubateur avec agitation
- Centrifugeuse pour microtubes avec température contrôlée
- Thermocycleur
Date de mise à jour : 22 août 2023